10月19日,我院生物物理团队黄胜友教授课题组在蛋白质-蛋白质相互作用预测领域取得重要进展,相关成果发表于《自然·机器智能》(Nature Machine Intelligence),题目为《基于几何三角感知和蛋白质语言模型的蛋白质-蛋白质接触预测》(Protein-protein contact prediction by geometric triangle-aware protein language models)。华中科技大学为唯一通讯单位,我院博士生林培聪为论文第一作者,博士生陶环宇和李豪为共同作者,黄胜友教授为通讯作者。

蛋白质通过与其他生物分子相互作用或者自组装形成复合物来发挥特定的功能。揭示蛋白质-蛋白质间的相互作用接触信息不仅能够帮助理解生命活动的分子机制,而且对于构建蛋白质复合物三维结构,进而研究其生物学功能有着重要的作用。随着人工智能方法的不断发展,蛋白质单体结构预测已经取得巨大的突破,尤其是AlphaFold2的问世,更是将单体结构预测的精度提升到实验级别。但是,蛋白质复合物结构预测仍然存在许多挑战,特别是异源复合物在预测精度仍有待提升。现有的蛋白质复合物链间接触预测方法存在着许多的不足。首先,大部分的方法只使用了卷积神经网络来提取局部特征,而蛋白质复合物中相互作用的氨基酸对在序列上存在许多长程相互作用,这需要更深的网络来提升感受野,其次,主流算法预测的蛋白质-蛋白质链间接触图中存在大量违背几何约束的接触对。
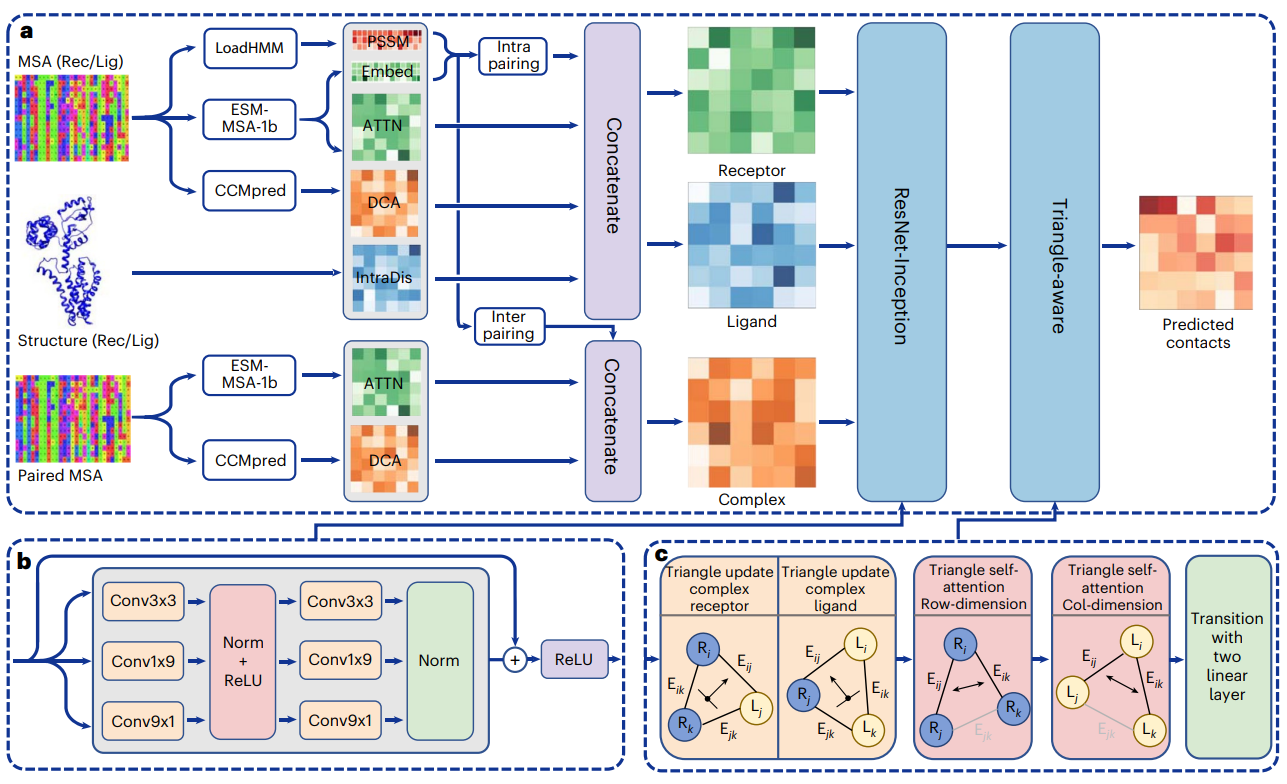
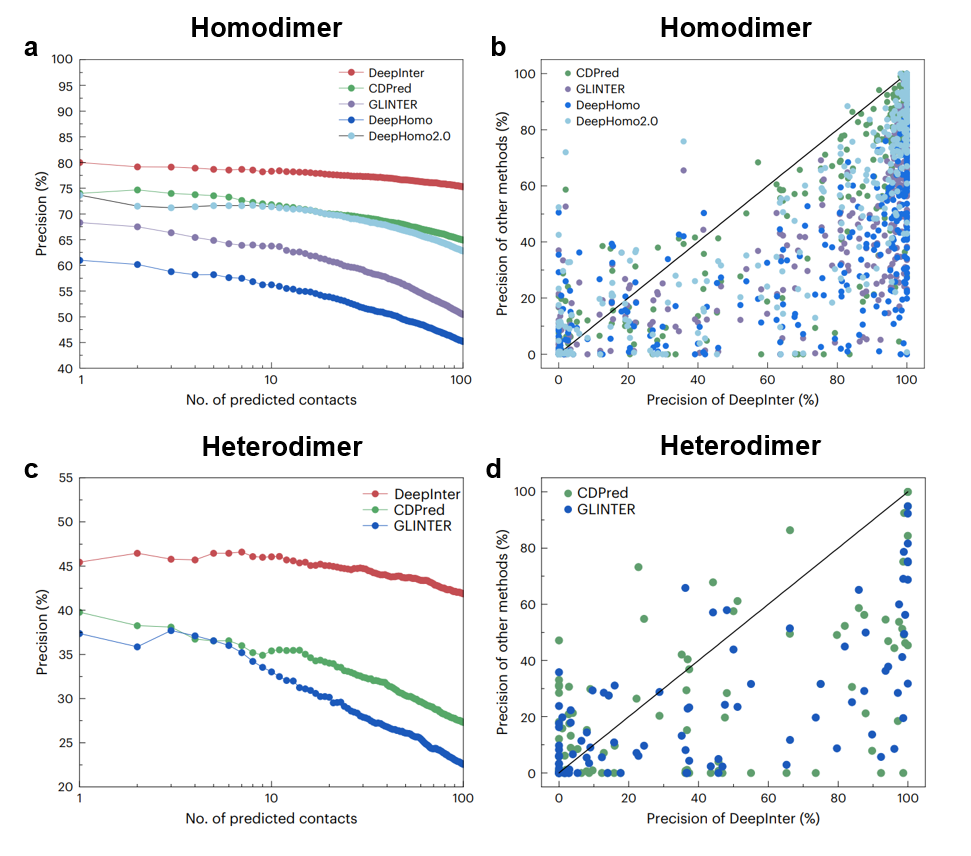
为了解决这一问题,黄胜友教授课题组提出了一种基于几何三角感知和蛋白质语言模型的蛋白质复合物链间接触预测方法-DeepInter。DeepInter设计了受体、配体、复合物三个分支路线的网络流程,以及独特的蛋白质间三角感知模块来有效提升二聚体复合物中链间接触预测的准确率和鲁棒性,同时能够减小预测的链间接触图中违背几何约束的数量。首先,DeepInter通过搜索受体和配体的多序列比对信息,并通过系统发育学的序列配对方法来构建复合物的多序列比对。其次,DeepInter利用先进的蛋白质语言模型分别提取了单体和复合物的序列表征和隐含的共进化模式。之后,DeeInter通过ResNet-Inception模块将输入特征编码到高维的潜空间,并通过Triangle-aware模块来同时考虑蛋白质内部和蛋白质之间的氨基酸对距离信息来驱使网络模型能够自动识别几何约束信息。通过在同源二聚体和异源二聚体的测试集对DeepInter进行全面的评估,并与其它主流的复合物接触预测算法进行比较。结果表明,DeepInter在不同的测试集上均展现出更高的准确率和鲁棒性。此外,随着考虑的预测链间接触对数量增加,DeepInter准确率相对其他方法来说更具稳定性(即下降曲率更低),说明我们提出的Triangle-aware模块能更好的捕捉全局相互作用,并减少其中的几何冲突。进一步地,DeepInter还在多序列深度,接触密度,构象变化,固有无序蛋白和结构相似度等多个影响因子上进行全面的分析和比较,表明我们方法相较其它方法具备更好的鲁棒性。
黄胜友教授课题组多年来一直致力于生物大分子(即蛋白质和核酸)相互作用计算及其复合物结构预测研究,在蛋白质-蛋白质、蛋白质-多肽、蛋白质-小分子、蛋白质-核酸相互作用及其复合物结构预测方面做了许多重要的工作,发展和开发了一系列方法、软件和计算平台,在NatureMachine Intelligence、Nature Protocols、Nature Communications、Journal of the American Chemical Society、Proceedings of National Academy of Sciences、Nucleic Acids Research等国际著名期刊发表多篇论文。
该项研究工作得到了国家自然科学基金(32161133002,62072199)和学校人才引进基金的资助。
论文链接:https://doi.org/10.1038/s42256-023-00741-2